16S rRNA高通量测序
 |
项目名称 | 16S rRNA 高通量测序 |
| 项目周期 | 根据实验量待定 | |
| 项目报价 | 280元/例 |
项目简介
宏基因组研究通常分析原核生物的16S 核糖体RNA基因(16S rRNA),16S 基因全长1500bp在保守区域中9V区),通常用可变区序列区分不同微生物的种属。使用哪个V区分析是有争议的,具体可以根据实验目的、设计和样本类型进行调整。这个方案分析的是16S V3和V4区,这个方案也可以分析其他V区的,只需要更换相应V区特异的引物即可,也可用于其他感兴趣的靶向扩增子测序。这个方案整合了桌面式的测序系统、机载初级和二级数据分析软件,机载的Local Run Manager软件或云端的BaseSpace Hub 提供完整的16S rRNA扩增子测序分析流程。
方案概述:
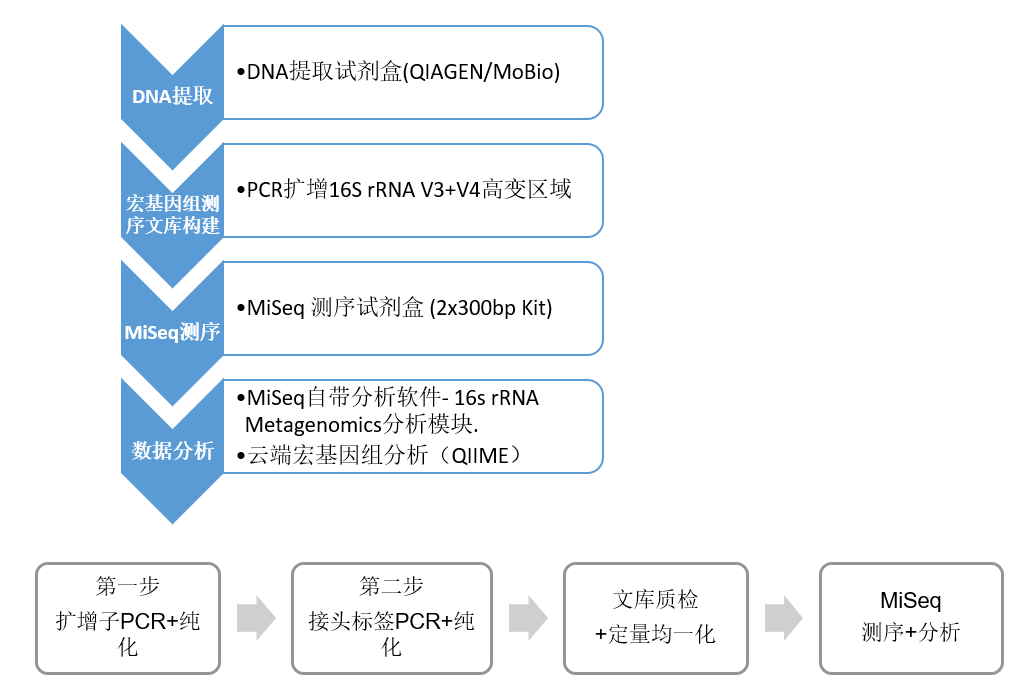
1.订购引物:扩增子全长大概460bp包含V3和V4两个可变区,引物两端带有与illumina接头和标签互补的序列,方便通过PCR直接实现文库的构建。Illumina不提供引物,引物需从第三方的公司合成订购。
2.构建文库:本方案包括了通过PCR扩增V3-V4区和通过PCR(很少几个cycle)加接头和双index标签的方法,用Nextera XT系列index可以最多将384个文库pooling在一起测序。
3.上机测序:在Miseq上测序用V3版试剂测序,双端300bp读长测序策略Read1、Read2部分区域重叠校准,可获得高质量全长的V3-V4序列。56小时可以完成一轮测序产出超过20M条序列,如果按一次上机96个样本计算,每个样本可获得远超过10万条序列/样本的数据量,充分满足通常宏基因组分析所需的数据量。
4.分析数据:机载软件或BaseSpace(云端)上有完整的宏基因组分析二级软件,用户可以选择使用Greengene数据库和RDP数据库分析统计,并以图形表格等形式展示属或种级别的微生物分类和数量。
报价说明
- 280元/例
客户提供
- 样品
品求
- 土壤,水体,口腔,粪便,淤泥等尽量提供新鲜的样品,如需保存,请收集样品后立刻放入-80℃冻存。
服务流程
结果交付
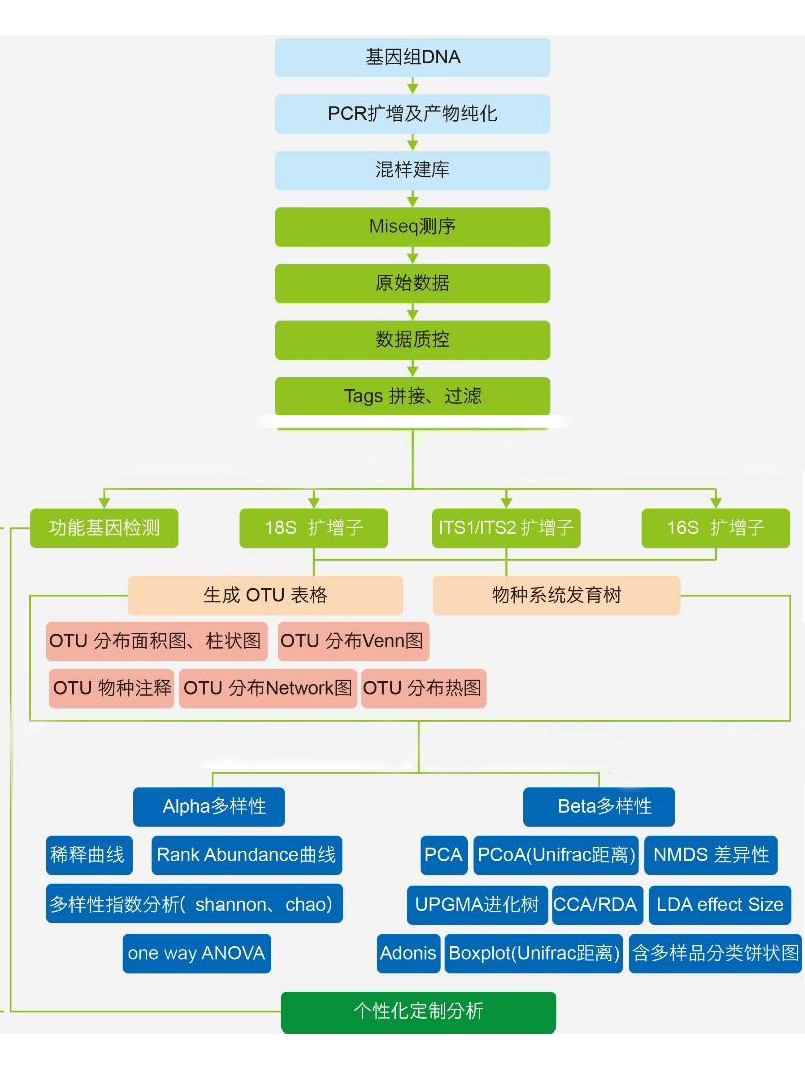
数据分析流程
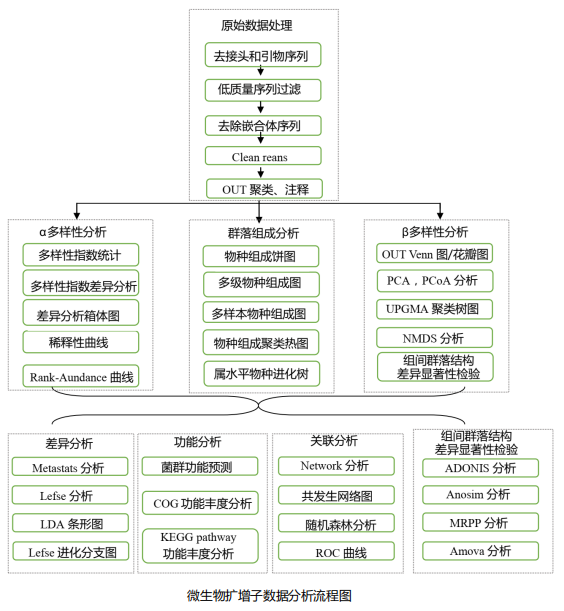
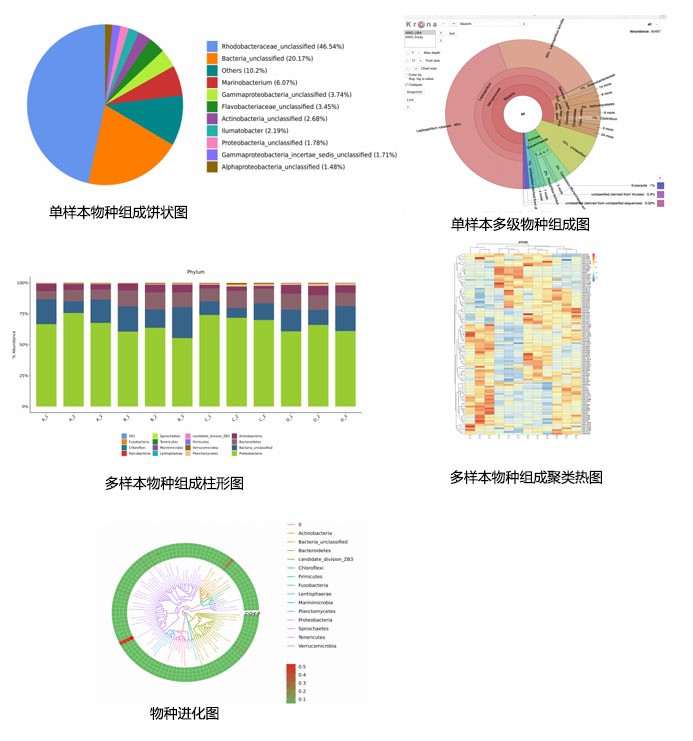
1. 群落组成分析
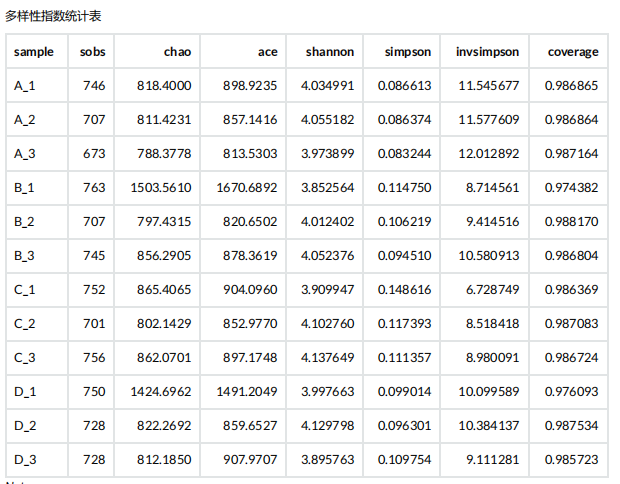
2. Alpha 多样性分析
Alpha多样性(Alpha diversity)是对单个样品中物种多样性的分析, 即分析 样本内(Within-community)的微生物群落多样性 ,通过单样本的多样性分析(Alpha 多样性)可以反映样本内的微生物群落的丰富度和多样性,包括用物种累积箱形图、物种多样性曲线和一系列统计学分析指数来评估各样本中微生物群落的物种丰富度和多样性的差异。



3. Beta 多样性分析
Beta多样性(Beta Diversity)是对不同样本的微生物群落构成进行比较分析, 即分析样本间的微生物群落多样性 。首先根据所有样本的物种注释结果和OTUs的丰度信息,将相同分类的OTUs息并理ProÖling Table)。同时利用OTUs之间的系统发生关系,进一步计算Unifrac距离(Unweighted Unifrac)。UniFrac是通过利用系统进化的信息来比较样品间的物种群落差异。 其计算结果可以作为一种衡量Beta Diversity的指数,它考虑了物种间的进化距离, 该指数越大表示样品间的差异越大 。然后,利用OTUs的丰度信息对Unifrac距离(非加权UniFrac , Unweighted Unifrac)进一步构建Weighted Unifrac距离。最后,通过多变量统计学方法主成分分析(PCA,Principal Component Analysis),主坐标分析(PCoA, Principal Co-ordinates Analysis),无度量多维标定法(NMDS,Non-Metric Multi- Dimensional Scaling),非加权组平均聚类分析(UPGMA,Unweighted Pair-group Method with Arithmetic Means)分析以及Beta多样性指数组间差异分析等方法,从中现不同样本(组)间的差异。



企业微信
|
|||||
| 了解详情,请联系孙琳琳:13194220518 | |||||


